微生物影响机制探究通常将核心讨论点聚焦于“物种”,总是倾向于找到某一个或几个物种与分组差异的关联,采用统计检验、LEfSe、随机森林、ROC、Indicator等各种指示物种研究方法。
但是细想,分组差异不一定是一个、几个物种的决定性作用,有可能是群落整体的影响。肠型分析就是这种站在群落整体角度,探究分组差异的研究思路,更侧重于分析样本群落特征(即肠型类别)与分组差异的关联。
“肠型”最初是学者在大样本量人肠道微生物研究过程中,对样本肠道微生物特征的分类[1],后来在很多研究中都被证实存在肠型[2],还发现肠型类型可能跟疾病等表型有一定的关联特征,可以辅助分析物种与宿主的关联机制。所以肠型鉴定在人、小鼠肠道微生物中有广泛应用,但后续也逐渐推广到其他动物研究中。
什么是肠型呢?一开始听到这个概念,可能都会有一个误区,以为肠型是对某个样本进行鉴定,判断该样本属于哪种肠型。但实际上,肠型分析是对所有样本进行聚类,把样本分成不同类别后,判断每个类别是否符合某种肠型,也有可能都不符合。目前主流的是基于数据库鉴定了三种肠型,也有研究提出四种肠型[2]。我们以三种肠型的分析方法为例和大家分享。
分析步骤
1)计算距离矩阵:最常用的是选择属水平相对丰度(也有文章选择科等其他水平),计算样本间Jensen-Shannon divergence(JSD)距离。
2)选择最佳cluster数目:计算Calinski–Harabasz(CH)index(图1),选择CH值最高时对应的K值,即为样本分类数(cluster的数量),通常在2到4之间比较好。
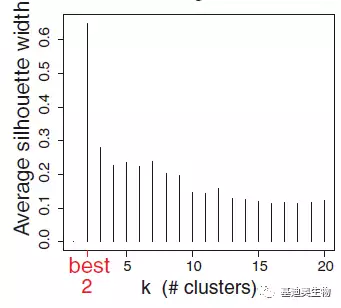
图1 CH index
3)样本聚类:基于Partitioning Around Medoids算法,R语言cluster包,将样本按预定数量进行分类,然后绘制PCoA等图形,按分类结果给定分组颜色(图2)。
4)样本聚类质量评估:计算样本silhouette value,值越高样本越符合聚类结果。
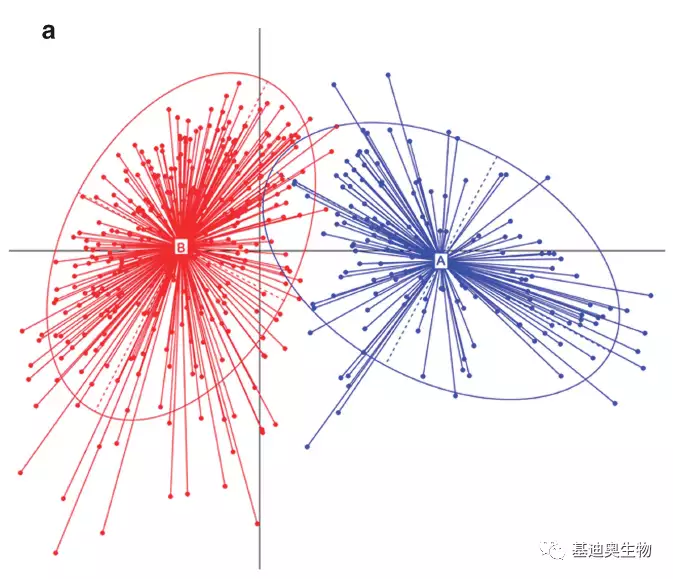
图2 样本聚类结果
肠型鉴定
以人、鼠为例,有3种肠型特征,Bacteroides(拟杆菌)是肠型1(enterotype 1,ET B)最好的指示类群;肠型2(enterotype 2,ET P)是由Prevotella(普氏菌) 驱动;肠型3(enterotype 3,ET F)是通过Firmicutes(厚壁菌)占比高低来区分的,其中最主要的类群为Ruminococcus(瘤胃球菌)。
肠型分类通常基于属水平进行,因为属水平可以更好的反映生态位变化。基于样本的聚类特征,设定分组,统计目标物种在组间的丰度差异,结合盒型图、小提琴图等可视化展示,基于肠型标志物种判断cluster的类型(图3)。

图3 标记物种丰度统计
肠型应用
对婴儿肠道微生物群的干扰会影响免疫系统的发育、过敏性疾病的风险。研究结合饮食类型,探讨肠道菌群在特应性皮炎(AD)患者中的作用。采集健康婴幼儿20例,AD患儿20例宏基因组测序[3]。如图4,样本最佳聚类分为2个cluster,绿色和红色,并使用盒型图展示红色cluster特征物种丰度,绿色cluster特征物种丰度。
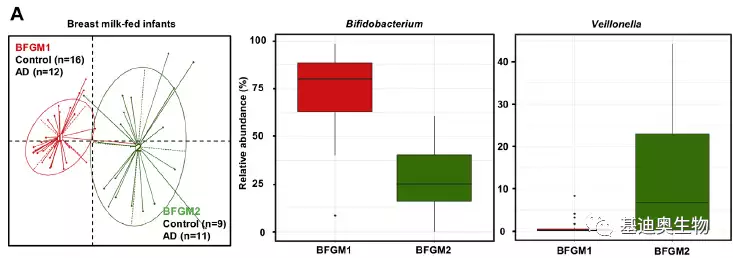
图4 肠型分析
Tips
基迪奥专业定制的微生物群落测序分析,如16S扩增子、宏基因组等,都可以提供个性化的肠型分析哦。有测序意向的小伙伴,欢迎咨询当地销售。
参考文献
[1] Arumugam, M., Raes, J., Pelletier, E., Le Paslier, D., Yamada, T., Mende, D. R., Fernandes, G. R., Tap, J., Bruls, T., Batto, J. M., Bertalan, M., Borruel, N., Casellas, F., Fernandez, L., Gautier, L., Hansen, T., Hattori, M., Hayashi, T., Kleerebezem, M., Kurokawa, K., … Bork, P. (2011). Enterotypes of the human gut microbiome. Nature, 473(7346), 174–180.
[2] Costea PI, Hildebrand F, Arumugam M, et al. Enterotypes in the landscape of gut microbial community composition [published correction appears in Nat Microbiol. 2018 Feb 13]. Nat Microbiol. 2018;3(1):8-16.
[3] Lee MJ, Kang MJ, Lee SY, et al. Perturbations of gut microbiome genes in infants with atopic dermatitis according to feeding type. J Allergy Clin Immunol. 2018;141(4):1310-1319.

